Идентификация и внутривидовое типирование возбудителей сапа и мелиоидоза 03. 00. 07 микробиология 03. 00. 15 генетика
| Вид материала | Автореферат |
СодержаниеB. mallei. Набор произвольных праймеров для дифференциации штаммов возбудителей сапа и мелиоидоза методом RAPD 23S рРНК, флагеллярного гена fliC |
- Контрольная работа по дисциплине Микробиология По теме Общая и специальная микробиология, 208.97kb.
- Рабочая программа генетика Код дисциплины по учебному плану опд ф. 6 для студентов, 274.5kb.
- Выделение и идентификация нетуберкулезных микобактерий у пациентов фтизиатрических, 706.97kb.
- Генетика Генетика, 18.18kb.
- Программа элективного курса по биологии для учащихся 10-х классов химико-биологического, 154.46kb.
- Разработка лабораторных экспресс-методов и технологии производства иммунодиагностических, 571.84kb.
- Темы: общая генетика, генетика человека, медицинская генетика. Вопросы для самоподготовки., 39.67kb.
- Примерная программа дисциплины микробиология, вирусология, микробиология полости рта, 447.35kb.
- Молекулярные механизмы формирования множественной лекарственной резистентности у Burkholderia, 787.44kb.
- Прототипы сибиреязвенных вакцин на основе генно-инженерных бациллярных штаммов и синтезируемых, 655.67kb.
Как следует из представленной таблицы, штаммы возбудителя мелиоидоза имели различные СТ (сиквенс-типы), что позволяло их дифференцировать друг от друга, в отличие от штаммов B. mallei, которые имели одинаковый 40 СT.
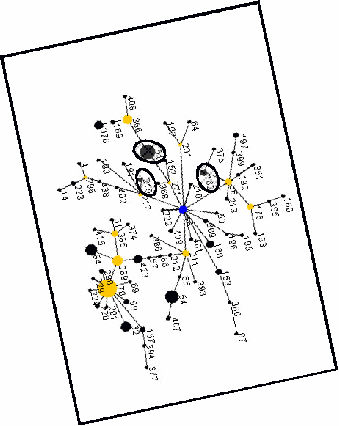
Использование этой технологии и алгоритма BURST (Based Upon Related Sequence Types) (Feil J.E. et al., 2004) позволило установить, что авирулентный штамм B.pseudomallei 107 с неизвестным источником выделения, который получен нами от A. Bremmelgaard и числящийся в ее коллекции как атипичный штамм возбудителя мелиоидоза (Bremmelgaard A., 1975), принадлежит, наряду с анализируемыми B. pseudomallei 100 и B. pseudomallei С-141 к клональным комплексам штаммов Юго-Восточной Азии (рис. 4).

B.pseudomallei 100

Рис. 4. Диаграмма различных СТ штаммов B.pseudomallei, выделенных в Юго-Восточной Азии (Алгоритм BURST). Стрелками отмечены СТ штаммов из коллекции ВолгоградНИПЧИ.
Полученные нами результаты и литературные данные свидетельствуют, с одной стороны, о значительно большей генетической однородности B. mallei (Godoy D. et al., 2003), а с другой, что метод МЛСТ недостаточно эффективен для внутривидового типирования возбудителя сапа и может быть использован лишь для дифференциации штаммов B. pseudomallei.
Оценка схемы типирования на основе амплификации вариабельных ампликонов (Variable Amplicon Typing Scheme), предложенной K. Duansonk с соавторами (2006), нами проведена на 18 штаммах возбудителя мелиоидоза и 4 штаммах возбудителя сапа из коллекции ВолгоградНИПЧИ.
В ходе эксперимента при анализе вариабельных продуктов амплификации (VAT), в зависимости от используемых праймеров и анализируемых штаммов, зарегистрировано от 3 до 9 фрагментов ДНК размерами от 132 до 788 п.н. Исследуемые 18 штаммов возбудителя мелиоидоза по наличию вариабельных ампликонов разделены на 13 VAT типов (рис.5). Большинство вариабельных локусов, выявленных с помощью субтрактивной гибридизации и на основе которых проводится типирование штаммов возбудителя мелиоидоза (Duangsonk K. et al., 2006), отсутствуют в геномах секвенированных штаммов B.mallei, поэтому нам не удалось разделить штаммы возбудителя сапа на различные генетические группы.

Рис. 5. Кластерный анализ VAT профилей штаммов возбудителя мелиоидоза.
Генетическая гетерогенность B.mallei была оценена нами методами рестрикционного анализа в формате пульс-электрофореза, VNTR-анализа и амплификации с произвольными праймерами, результаты которых позволили провести эффективную внутривидовую дифференциацию штаммов возбудителя сапа.
С помощью ПЭФ все исследуемые 14 штаммов возбудителя сапа разделены на 13 кластерных групп (90 % сходства). На уровне 92 % сходства каждый штамм B.mallei формировал уникальную кластерную группу (рис. 6).
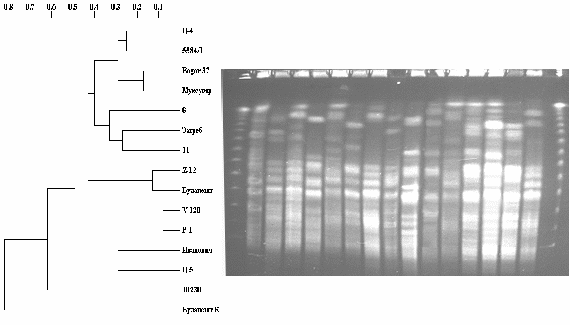
Рис. 6. Электрофореграмма Xba I рестрикционных фрагментов ДНК и дендрограмма сходства штаммов B. mallei. ПЭФ в 1% агарозном геле с окраской ДНК этидиумом бромидом.
Амплификация с произвольными праймерами (метод RAPD) является одним из наиболее простых способов дифференциации штаммов. Данный метод использован для многих микроорганизмов, в том числе B.pseudomallei (Haase A. et al., 1995; Leelayuwat C. et al., 2000). Для генотипирования штаммов B.mallei этот методический прием не применяли.
Практически во всех цитируемых работах отмечено, что метод амплификации с произвольными праймерами требует не только строгой стандартизации подготовки культур, но и условий проведения ПЦР и электрофореза. Для получения высокоинформативных и воспроизводимых ДНК-профилей нами первоначально оптимизированы параметры реакции амплификации - отработаны температурные режимы ПЦР, определены необходимые концентрации праймеров, исследуемой ДНК, Taq-полимеразы, а также режимы электрофоретического разделения фрагментов амплификации.
Детекцию фрагментов, как и в случае ПЭФ, осуществляли в автоматическом режиме программы RFLPscan 3.12 из пакета программ Gene Profiler 4.03 (CSP Inc., USA). Кластерный анализ и графическое отображение матриц коэффициентов сходства проводили при помощи программы TreeCon for Windows v.1.3b. Группирование штаммов и построение дендрограмм проводили при помощи невзвешенного парно-группового метода UPGMA (Sneath P.H., Sokal R.R., 1973).
Из 19 проанализированных нами произвольных олигонуклеотидных затравок, для дифференциации штаммов патогенных буркхольдерий экспериментально подобран набор праймеров, показавших наибольшую эффективность (табл. 3). Каждые из олигонуклеотидов или их комбинаций давали возможность обнаружить специфические микролокусы в геномах штаммов, которым свойственна различная степень нуклеотидной изменчивости на внутривидовом уровне.
Таблица 3
Набор произвольных праймеров для дифференциации штаммов возбудителей сапа и мелиоидоза методом RAPD
| № п/р | Обозначение | Последовательность | Автор |
| 1 | RA | GTTTCGCTCC | (Wongratanacheewin S., 2000) |
| 2 | Jeffreys | GGAGGTGGGCAGGAXG | (Jeffreys A.J., 1985) |
| 3 | Rep | CGACGCAGGC | (Liu Y., 2002) |
| 4 | PR 6 | GAGACGCACA | (Haase A., 1995) |
| 5 | PR 7 | CAGCCCAGAG | (Haase A., 1995) |
| 6 | PR 13 | AGCGTCACTC | (Haase A., 1995) |
| 7 | PR 16 | AAGCGACCTG | (Haase A., 1995) |
При анализе электрофоретических профилей ДНК 14 штаммов возбудителя сапа выявлены схожие RAPD-паттерны, которые, независимо от используемых праймеров, имели коэффициент подобия более 75 %, что доказывало высокую степень однородности изучаемых штаммов B.mallei. Анализ электрофоретических RAPD - паттернов штаммов B.pseudomallei и B.thailandensis показал их более высокую полиморфность по сравнению со штаммами возбудителя сапа. Данная закономерность вполне объяснима с позиций формирования адаптационной и генетической изменчивости у этой триады буркхольдерий. Кроме того, размер геномов B. pseudomallei и B.thailandensis превышает размер генома B. mallei, что также может влиять на количество RAPD-фрагментов на электрофореграммах.
Нами выявлено, что практически любой из праймеров пригоден для видовой дифференциации патогенных буркхольдерий. Однако в зависимости от праймеров формировалось различное количество и состав кластерных групп. Образованные кластерные группы отражали гетерогенность популяций патогенных буркхольдерий, при которой каждый штамм имел свой характерный RAPD-тип. Анализ штаммов, проведенный внутри каждого кластера в отдельности, показал, что при использовании разных праймеров формируются независимые группы.
Для проведения внутривидового типирования штаммов возбудителя сапа наиболее эффективны оказались праймеры Jeffreys, PR 6, PR 7 и RA, в то время как для для возбудителя мелиоидоза – олигонуклеотидные затравки PR 7, PR 16, PR 13, RA и Rep. При суммировании результатов RAPD-типирования и формировании единой матрицы сходства с использованием всего набора исследуемых праймеров каждый штамм B.mallei и B.pseudomallei формировал уникальную кластерную группу, соответственно количеству исследуемых штаммов, отражая гетерогенность популяций возбудителей сапа и мелиоидоза (рис.7).
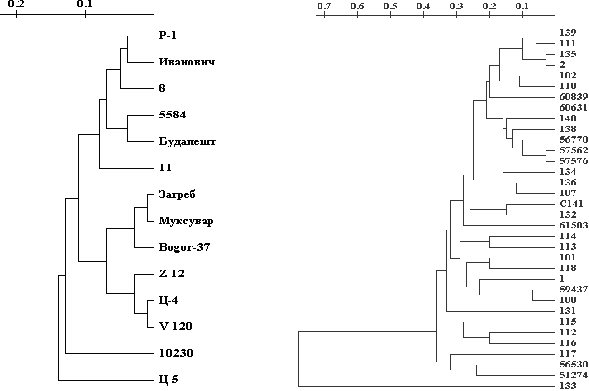
Рис. 7. Дендрограмма сходства штаммов патогенных буркхольдерий с использованием набора произвольных праймеров
Детальный анализ геномов различных микроорганизмов, в том числе B. pseudomallei и B. mallei позволил обнаружить многочисленные повторяющиеся последовательности. Одними из них являются вариабельные тандемные повторы, лежащие в основе VNTR-анализа (Keim P. et al., 2000; Farlow J. et al., 2001; Сучков И. Ю. с соавт., 2004; Цыганкова О. И. с соавт., 2006). У патогенных буркхольдерий впервые тандемный повтор размером 10 п.н., обнаружен при анализе штамма B.pseudomallei ATCC 23343 (Liu Y. et al., 2002).
В настоящем разделе работы была рассмотрена возможность использования VNTR-анализа для генотипирования коллекционных штаммов патогенных буркхольдерий. Амплификацию VNTR-локусов возбудителей сапа и мелиоидоза проводили с помощью праймеров SR1/SR5 (Liu Y. et al., 2002).
В результате амплификации во всех исследуемых штаммах возбудителей сапа и мелиоидоза синтезировались фрагменты ДНК размером от 422 до 692 п.н. (рис. 8).
1
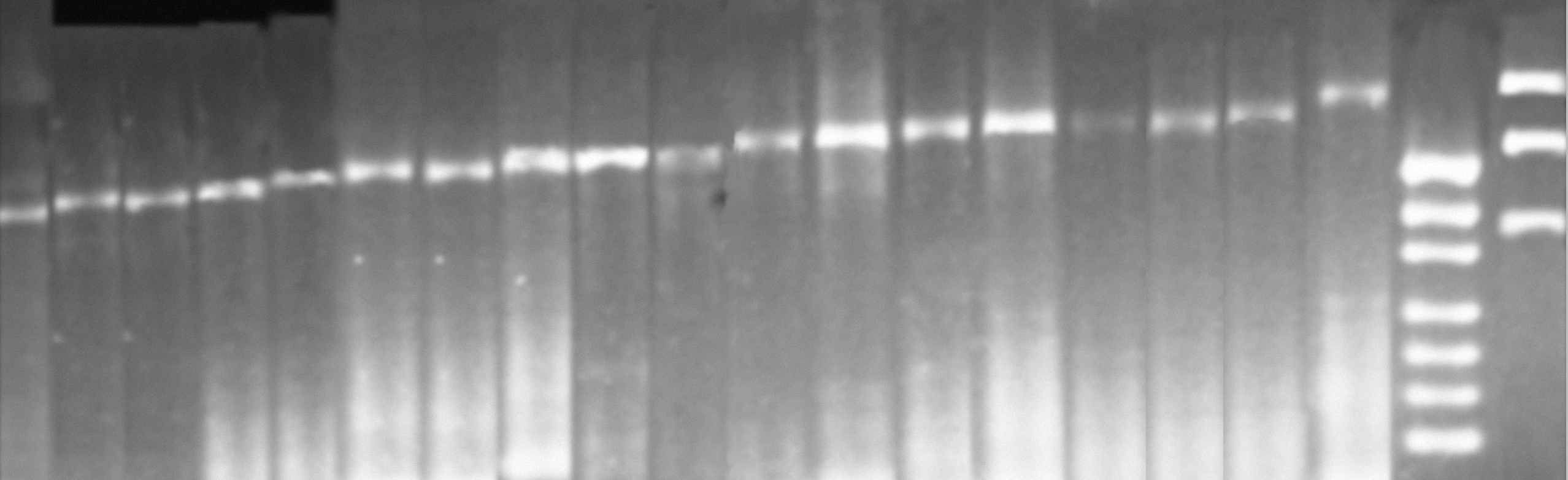 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20
2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20Рис. 8. Электрофореграммы продуктов амплификации штаммов патогенных буркхольдерий с праймерами SR1/SR5.

На основе различий в размерах полученных ампликонов при анализе 6 штаммов В.pseudomallei и 11 штаммов В. mallei нами идентифицировано 11 аллелей (VNTR-типов). Шесть штаммов B.pseudomallei разделены на 5 аллелей. Среди 11-ти штаммов B.mallei идентифицировано 8 аллелей с различным числом тандемных повторов. Причем, два аллеля были общими для B.mallei и B.pseudomallei.
Стабильность тандемных повторов для каждого штамма и отсутствие изменений нуклеотидных последовательностей в этом генетическом локусе после пассажей на животных, как отмечено в работе Y. Liu с соавторами (2002), свидетельствует о возможности использования данного генетического маркера в VNTR анализе B.mallei и B.pseudomallei. При этом, амплификационная тест-система с праймерами SR1/SR2 пригодна для простого, быстрого и высокоспецифичного ПЦР-типирования штаммов патогенных буркхольдерий.
Обобщая полученные результаты необходимо отметить, что в основу алгоритма генодиагностики и генотипирования патогенных буркхольдерий нами был заложен принцип дифференцированного подхода к выбору наиболее информативного метода или сочетаний молекулярно-генетических методов. Это позволило разработать единую схему идентификации и типирования B.mallei и B.pseudomallei (рис.9).
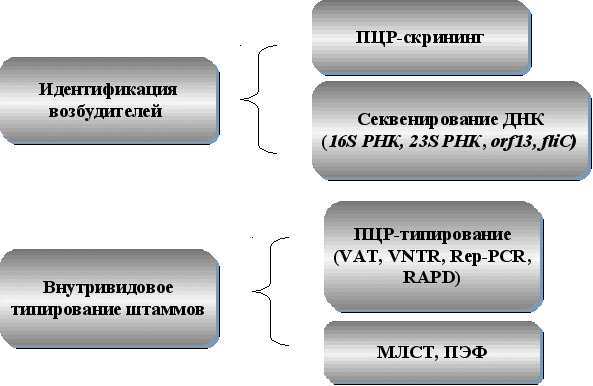
Рис. 9. Схема генетической идентификации и типирования B.mallei и B.pseudomallei.
В данной схеме ПЦР-скрининг предполагает использование амплификационных тест-систем со специфическими праймерами bfl1s/bfl2a, BTTS-s1/BTTS-as3 и b23s7/b23a8 для детекции B.mallei и B.pseudomallei. Алгоритм генодиагностики и генотипирования патогенных буркхольдерий, включает на этапе идентификации культур реакцию амплификации и секвенирование ампликонов. Для дифференциации штаммов возбудителя мелиоидоза пригодны все используемые в работе генетические методы. Однако для ускоренного генотипирования B.pseudomallei целесообразно использовать различные варианты ПЦР-типирования (VAT, RAPD, Rep-ПЦР, VNTR). К этому следует добавить, что для дифференциации штаммов B.mallei эффективны ПЭФ, VNTR и RAPD, но быстрое генетическое типирование штаммов возбудителя сапа обеспечивают метод анализа тандемных повторов и амплификация с произвольными праймерами.
Выводы
- Оценена сравнительная эффективность молекулярно-биологических методов идентификации и внутривидовой дифференциации штаммов B. mallei и B. pseudomallei. Предложена схема лабораторной диагностики сапа и мелиоидоза с включением ПЦР как на этапе идентификации чистых культур, так и анализа проб клинического материала и объектов внешней среды. Сформирована единая система генодиагностики и генотипирования патогенных буркхольдерий.
- На основе нуклеотидных последовательностей генов, детерминирующих 23S рРНК, флагеллярного гена fliC и orf гена III типа секреции, определены олигонуклеотидные праймеры, подобраны оптимальные условия, определены параметры проведения ПЦР и сконструированы амплификационные тест-системы для идентификации патогенных буркхольдерий, позволяющие выявлять возбудителей сапа и мелиоидоза в концентрациях 1х102 -1х103 м.к./мл.
- Показано, что использование олигонуклеотидных праймеров burts1/burta2, сконструированных на основе флагеллярного гена fliC, позволяет идентифицировать три вида буркхольдерий B.pseudomallei, B.mallei и B.thailandensis при анализе чистых культур и обнаруживать B.thailandensis в пробах экспериментального клинического материала при моделировании инфекционного процесса.
- Для повышения специфичности идентификационных тестов необходимо использовать ПЦР с диагностическими праймерами, сконструированными на различные ДНК-мишени. Показано, что сочетание реакции амплификации и секвенирование ДНК мишеней - участков 16S рРНК и 23S рРНК, orf13 или fliC является алгоритмом быстрой идентификации B. mallei и B. pseudomallei и их дифференциации от близкородственных видов микроорганизмов.
- Установлено, что эффективными фенотипическими методами дифференциации штаммов возбудителя мелиоидоза являются фаготипирование и анализ белкового спектров, в то время как проведение внутривидового типирования штаммов B.mallei возможно на основе различий в спектре суммарных клеточных белков. Тесты, основанные на выявлении вариабельных биохимических признаков и различий в чувствительности к антибиотикам выступают лишь в качестве дополнительных критериев внутривидовой дифференциации штаммов патогенных буркхольдерий.
- На основе плазмидного анализа выявлено 4 группы штаммов В.pseudomallei, отличающихся по составу и размеру плазмидных репликонов. Показано, что обнаруженные плазмиды могут выступать в качестве дополнительного критерия генотипической дифференциации штаммов и являться эпидемиологическим маркером в случае возникновении инфекционного процесса, вызванного плазмидсодержащим штаммом B.pseudomallei.
- Показана высокая эффективность метода мультилокусного сиквенс-типирования для дифференциации штаммов возбудителя мелиоидоза, позволившего установить, что анализируемые штаммы B.pseudomallei 100, С-141, а также B.pseudomallei 107 с неизвестным источником выделения, относятся к Юго-Восточной группе. Все исследуемые штаммы B. mallei формируют единый 40 сиквенс-тип, входящий в клональный комплекс штаммов B. pseudomallei.
- Отработаны условия проведения рестрикционного анализа ДНК штаммов возбудителя сапа, оценена их генетическая гетерогенность и показана высокая эффективность метода пульс-электрофореза для внутривидового генетического типирования штаммов B.mallei. На основе полиморфизма длин рестрикционных фрагментов 14 штаммов возбудителя сапа с помошью кластерного анализа разделены на 13 генетических групп.
- Показана высокая эффективность RAPD-типирования для дифференциации штаммов патогенных буркхольдерий. Предложен набор олигонуклеотидных затравок, обозначенных Jeffreys, PR 6, PR 7 и RA для проведения внутривидового типирования штаммов возбудителя сапа и праймеров PR 7, PR 16, PR 13, и RA – для возбудителя мелиоидоза.
- Выявлена эффективность схемы типирования, основанной на анализе вариабельных ампликонов, для ускоренной генетической дифференциации штаммов возбудителя мелиоидоза, в отличие от штаммов B.mallei, в геномах которых вариабельные ампликоны не выявлены. С помощью ПЦР-типирования 18 штаммов возбудителя мелиоидоза разделены на 13 генетических группп (VAT типов).
- Установлена пригодность праймеров, фланкирующих тандемные повторы в гене, детерминирующем эстеразу патогенных буркхольдерий, для простого, быстрого и высокоспецифичного ПЦР-типирования штаммов возбудителей сапа и мелиоидоза. С помощью VNTR анализа среди 11 штаммов B.mallei идентифицировано 8 аллелей с различным числом тандемных повторов, в то время как шесть штаммов B.pseudomallei разделены на 5 аллелей.
- Показано, что алгоритм быстрой дифференциации штаммов B.pseudomallei включает методы ПЦР-типирования, основанные на анализе вариабельных ампликонов и количества тандемных повторов, амплификации ДНК с произвольными праймерами и Rep-ПЦР, в то время как быстрое генетическое типирование штаммов B.mallei обеспечивают VNTR-анализ и амплификация с произвольными праймерами.
Список работ, опубликованных по теме диссертации
- Генетика возбудителя мелиоидоза / Меринова Л.К., Гришкина Т.А., Тарасова Т.Д., Антонов В.А. // Мелиоидоз (под ред. акад. Н.Г.Тихонова).- 1995.- Волгоград.- С. 26-47.
- Антонов В.А., Замараев В.С., Захарова И.Б. Плазмиды возбудителя мелиоидоза // Экологоэпидемический надзор за природно-очаговыми инфекциями в Северном Прикаспии: Тез. науч. конф., посвящ. 95-летию Астраханской ПЧС, Астрахань, 1996.- С.107.
- Антонов В.А., Замараев В.С., Захарова И.Б. Изучение гомологии плазмид возбудителя мелиоидоза // Там же.- С.103.
- Антонов В.А., Замараев В.С., Меринова Л.К. Мобилизация криптических плазмид Pseudomonas pseudomallei в гетерологичные виды микроорганизмов // Там же.- С.99.
- Денисов И.И., Андрус В.Н., Антонов В.А. Адаптация псевдомонад, имеющих клиническое значение к дезинфектантам // Там же.- С.101.
- Илюхин В.И., Антонов В.А., Гришкина Т.А. Таксономическая позиция патогенных псевдомонад // Мат. VII съезда Всероссийского общества эпидемиологов, микробиологов и паразитологов, 28-31 января 1997, Москва. - 1997.- С.201-202.
- Антонов В.А., Меринова Л.К., Замараев В.С, Викторов Д.В. Анализ фенотипических свойств штаммов возбудителя сапа, несущих криптические плазмиды Pseudomonas pseudomallei // Мат. науч.-практ. конф., посвящ. 100-летию образования противочумной службы России, 16-18 сентября 1997, Саратов, 1997.- C.6-7.
- Замараев В.С., Антонов В.А. Плазмиды Pseudomonas pseudomallei // Мат. науч.-практ. конф., посвящ. 100-летию образования противочумной службы России, 16-18 сентября 1997, Саратов, 1997.- C.49-50.
- Антонов В.А., Меринова Л.К., Замараев В.С., Агеева Н.П., Викторов Д.В. Фенотипические свойства возбудителей сапа и мелиоидоза, содержащих плазмидные репликоны // Проблемы санэпид охраны территории стран СНГ: Тез. науч. практич. конф., 15-17 сентября, 1998.- Саратов, 1998.- С.174-176.
- Антонов В.А., Замараев В.С., Илюхин В.И. Группоспецифический ДНК-зонд для идентификации патогенных псевдомонад // Биотехнология .- 1998.-N5.- С.75-84.
- Антонов В..А., Меринова Л.К., Викторов Д.В., Замараев В.С., Захарова И.Б., Ткаченко Г.А. Влияние R-плазмид на отдельные фенотипические признаки штаммов возбудителя сапа и мелиоидоза // Диагностика, лечение и профилактика опасных инфекционных заболеваний. Биотехнология.Ветеринария: Мат. юбилейной науч. конф., посвящ. 70-летию НИИ микробиологии МО РФ, 30 ноября -1 декабря 1998, Киров, 1998.- C.48-49.
- Захарова И.Б., Замараев В.С., Антонов В.А., Ткаченко Г.А. Конструирование интегративного вектора на основе ColE1-репликона для клонирования в Burkholderia pseudomallei // Там же.- C.48-49.
- Замараев В.С., Антонов В.А., Илюхин В.И., Захарова И.Б. Выделение и первичная характеристика плазмид Pseudomonas (Burkholderia) pseudomallei // Мол. генет. микробиол. вирусол.- 1998.- N4.- С.28-33.
- Vicktorov D.V., Piven N.N., Zakharova I.B., Plekhanova N.G., Antonov V.A. Melioidosis vaccine: some approaches to development / First Global Conference on Vaccines and Immunisation into the next millennium.6th-9 th September 1999, Manchester, UK, 1999, р.47-48
- Антонов В.А., Замараев В.С., Меринова Л.К., Илюхин В.И. Burkholderia mallei как хозяин плазмид возбудителя мелиоидоза // Scientific Journal.- 1999.- №7.- р.225-226.
- Антонов В.А., Замараев В.С., Меринова Л.К., Захарова И.Б., Ткаченко Г.А, Бахтояров Г.Н., Клонирование фрагментов криптической плазмиды возбудителя мелиоидоза // Проблемы медицинской и экологической биотехнологии: Тез. докл. юбилейной науч. конф., посвящ. 25-летию ГНЦ прикладной микробиологии, 14-15 декабря 1999, Оболенск, 1999.- С.8.
- Антонов В.А., Меринова Л.К., Викторов Д.В., Ткаченко Г.А., Бахтояров Г.Н. Влияние плазмиды RP1::Tn10 на чувствительность клеток сапного микроба к мелиоидозному бактериофагу // Там же.- С.9.
- Захарова И.Б., Замараев В.С., Антонов В.А. Физическое картирование криптической плазмиды рРМ1 возбудителя мелиоидоза // Проблемы особо опасных инфекций.- 1999.- Вып.79.- С.159-162.
- Ткаченко Г.А., Антонов В.А., Бахтояров Г.Н. Плазмиды возбудителя мелиоидоза // Тез. конф. молодых ученых Поволжья и Северного Кавказа, 15-17 февраля, 2000.- Нижний Новгород, 2000.- С. 81.
- Меринова Л.К., Антонов В.А., Замараев В.С., Викторов Д.В. Мобилизация криптических плазмид возбудителя мелиоидоза в гетерологичные виды микроорганизмов // Мол. генет. микробиол. вирусол.- 2000.- N2.- С.37-40.
- Антонов В.А., Илюхин В.И., Замараев В.С., Трушкина М.Н. Использование плазмид возбудителя мелиоидоза в качестве ДНК-зондов для идентификации близкородственных микроорганизмов // Биотехнология .- 2000.-N4.- С.8-14.
- Антонов В.А., Замараев В.С., Захарова И.Б., Меринова Л.К., Викторов Д.В., Попов С.Ф. Изучение плазмид патогенных буркхолдерий // Природно-очаговые инфекции в Нижнем Поволжье: Сб.науч. тр. (под ред. Н.Г. Тихонова).- Волгоград: Изд-во «Принт», ВолГУ, 2000.- С.41-46.
- Илюхин В.И., Кисличкин Н.Н., Тихонов Н.Г., Плеханова Н.Г., Денисов И.И., Кисличкина И., Антонов В.А., Пивень Н.Н., Викторов Д.В., Фарбер С.М. Перспективы применения
